一文读懂 UniProt 数据库(2023 最新版)
| 阿里云国内75折 回扣 微信号:monov8 |
| 阿里云国际,腾讯云国际,低至75折。AWS 93折 免费开户实名账号 代冲值 优惠多多 微信号:monov8 飞机:@monov6 |

一、UniProt 数据库介绍
Uniprot Universal Protein 是包含蛋白质序列功能信息研究论文索引的蛋白质数据库整合了包括EBI European Bioinformatics InstituteSIBthe Swiss Institute of BioinformaticsPIRProtein Information Resource三大数据库的资源。
- EBI European Bioinformatics Institute欧洲生物信息学研究所EMBL-EBI是欧洲生命科学旗舰实验室EMBL的一部分。位于英国剑桥欣克斯顿的惠康基因组校园内是世界上基因组学领域最强的地带之一。
- SIBthe Swiss Institute of Bioinformatics瑞士日内瓦的SIB维护着ExPASy专家蛋白质分析系统服务器这里包含有蛋白质组学工具和数据库的主要资源。
- PIRProtein Information ResourcePIR由美国国家生物医学研究基金会NBRF于1984年成立旨在协助研究人员识别和解释蛋白质序列信息。
二、UniProt 数据库构成

目前UniProt由主要由以下子库构成
| 数据库名 | 全名 | 用途 |
|---|---|---|
| Swiss-Prot | Protein knowledgebas (review) | 高质量的、手工注释的、非冗余的数据库 |
| TrEMBL | Protein knowledgebase (unreview) | 自动翻译蛋白质序列预测序列未验证的数据库 |
| UniParc | Sequence | 非冗余蛋白质序列数据库 |
| UniRef | Sequence clusters | 聚类序列减小数据库加快搜索的速度 |
| Proteomes | Protein sets from fully sequenced genomes | 为全测序基因组物种提供蛋白质组信息 |
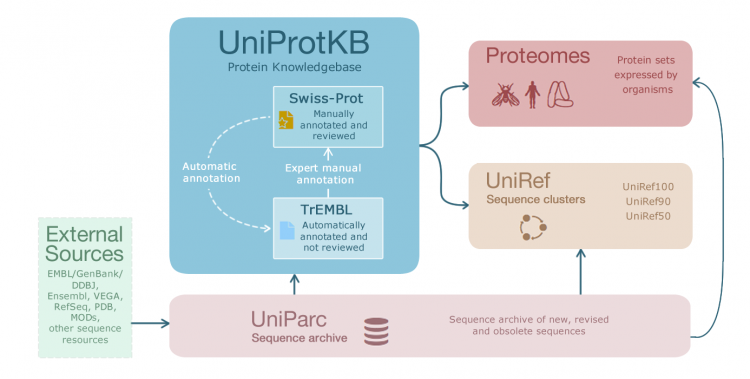
他们的关系如下
通过EMBLGenBankDDBJ等公共数据库得到原始数据处理后存入UniParc的非冗余蛋白质序列数据库。
UniParc作为数据仓库再分别给UniProtKBProteomesUniRef提供可靠的数据集。
这里的UniProtKB 由两个子库构成 Swiss-ProtTrEMBL。
Swiss-Prot 经过人工验证和注释是高质量的蛋白质注释数据。但人工效率在高速增长的蛋白质数据面前显得极其低效。因此注释这些数据需要大量时间为了弥补这一问题。
TrEMBL 被建立用于存储由机器自动翻译和预测的蛋白质序列。显然这必然导致蛋白质质量下降。
简而言之在UniProtKB数据库中Swiss-Prot是由TrEMBL经过手动注释后得到的高质量非冗余数据库也是我们今后常用的蛋白质数据库之一。
三、Swiss-Prot 介绍
高质量的、手工注释的、非冗余的数据集
Swiss-Prot 提供高水平注释例如蛋白质功能其域结构翻译后修饰变体等的描述和蛋白质序列。
Swiss-Prot由Amos Bairoch博士在1986年创建由瑞士生物信息学研究所开发随后由欧洲生物信息学研究所的Rolf Apweiler开发。也就是说EBI和SIB共同制作了Swiss-Prot和TrEMBL数据库。
注释主要来自文献中的研究成果和E-value校验过计算分析结果有质量保证的数据才被加入该数据库 。
手动注释
注释会用相关出版物通过搜索数据库例如PubMed进行识别。阅读每篇论文的全文然后提取信息并将其添加到条目中。科学文献中的注释包括但不限于
- 蛋白质和基因名称
- 功能
- 特定于酶的信息例如催化活性辅因子和催化残基
- 亚细胞定位
- 蛋白质相互作用
- 表达方式
- 重要域和站点的位置和角色
- 离子底物和辅因子结合位点
- 通过自然遗传变异RNA编辑替代剪接蛋白水解加工和翻译后修饰产生的蛋白质变异形式
计算机预测
Swiss-Prot条目的注释中使用了一系列序列分析工具包括手动检测和评估计算机预测。
这些预测包括翻译后修饰跨膜结构域和拓扑信号肽结构域识别和蛋白质家族分类。
也包括序列之间的差异可变剪接自然变异错误的起始位点错误的外显子边界移码未识别的冲突。
这里包含了很多第三方算法和软件
3.1、查询蛋白质基础操作
1、进入官网https://www.uniprot.org/

1、切换数据库也就是上面介绍的
2、输入基因名uniprot id物种名等都可以
3、点击进行搜索
4、进入搜索结果页面
https://www.uniprot.org/uniprotkb?query=oct4
新版界面主要由搜索框搜索结果表和左边过滤选项面板构成
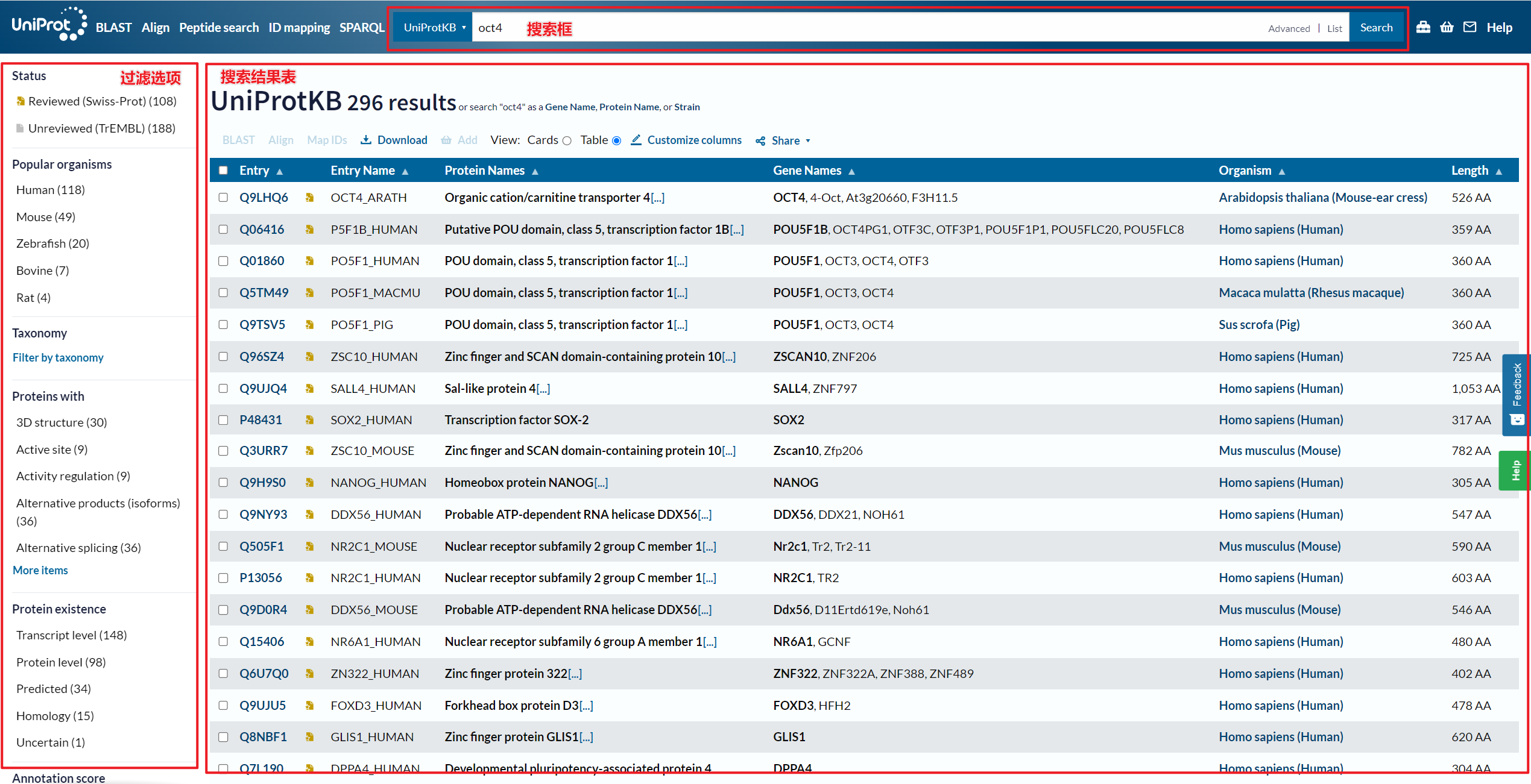
5、过滤选项面板
- 来源库Reviewed存储在Swiss-Prot数据库中经过验证的蛋白数据Unreviewed存储在TrEMBL数据库中没有经过验证的蛋白数据
- 物种点击
Popular organisms切换到该物种或者在Taxonomy通过 taxid 来筛选。什么是 Taxonomyhttps://blog.csdn.net/u011262253/article/details/95304930/ - 蛋白质相关注释内容直接点击对应管关键字
- 注释分数分数越高注释内容质量越高
- 蛋白质长度单位是氨基酸AA
6、搜索结果表
依次是Unprot ID蛋白质Uniprot名称蛋白质名称基因名物种名序列长
7、比对操作
- 序列与蛋白库比对如果需要Blast来查看某个蛋白有哪些序列相似的蛋白序列先选中感兴趣蛋白前的方框点击Blast
- 多序列比对先选中感兴趣蛋白前的方框点击Align
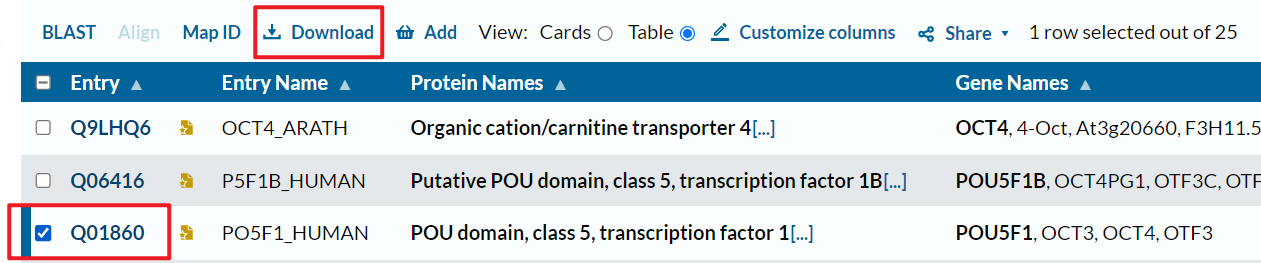
8、数据下载
下面以 PO5F1_HUMAN 为例。
先选中感兴趣蛋白前的方框点击Download下载。
下载对应的fasta序列来看看
3.2、UniProt Fasta 文件介绍
>sp|Q01860|PO5F1_HUMAN POU domain, class 5, transcription factor 1 OS=Homo sapiens OX=9606 GN=POU5F1 PE=1 SV=1
MAGHLASDFAFSPPPGGGGDGPGGPEPGWVDPRTWLSFQGPPGGPGIGPGVGPGSEVWGI
PPCPPPYEFCGGMAYCGPQVGVGLVPQGGLETSQPEGEAGVGVESNSDGASPEPCTVTPG
AVKLEKEKLEQNPEESQDIKALQKELEQFAKLLKQKRITLGYTQADVGLTLGVLFGKVFS
QTTICRFEALQLSFKNMCKLRPLLQKWVEEADNNENLQEICKAETLVQARKRKRTSIENR
VRGNLENLFLQCPKPTLQQISHIAQQLGLEKDVVRVWFCNRRQKGKRSSSDYAQREDFEA
AGSPFSGGPVSFPLAPGPHFGTPGYGSPHFTALYSSVPFPEGEAFPPVSVTTLGSPMHSN
首先看 > 后的注释信息
-
spSwiss-Prot数据库的简称也就是上面说的验证后的蛋白数据库
-
Q01860UniProt ID号
-
PO5F1_HUMAN是UniProt 的登录名
-
POU domain, class 5, transcription factor 1蛋白质名称
-
OS=Homo sapiensOS是Organism简称Homo sapiens为人的拉丁文分类命名也就是这是人的蛋白质
-
OX=9606Organism Taxonomy也就是物种分类数据库Taxonomy ID
-
GN=POU5F1Gene name基因名为POU5F1
-
PE=1Protein Existence蛋白质可靠性对应5个数字数字越小越可靠
-
- 1Experimental evidence at protein level
- 2Experimental evidence at tranlevel
- 3Protein inferred from homology
- 4Protein predicted
- 5Protein uncertain
-
SV=1Sequence Version序列版本号
3.3、查询蛋白质高级搜索
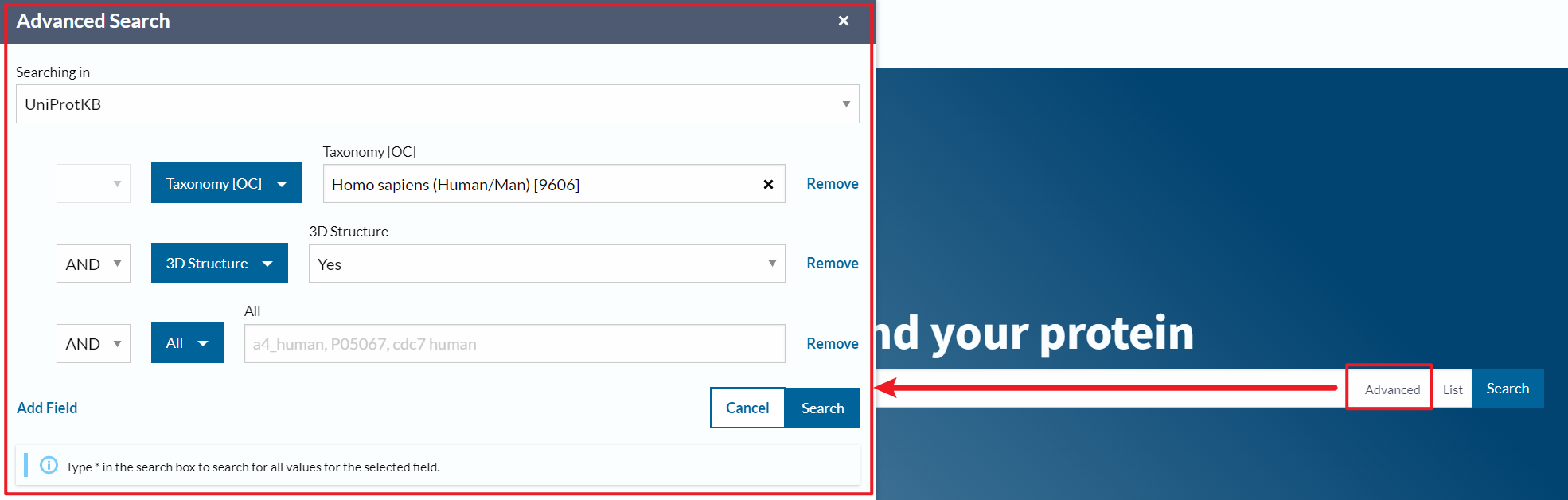
1、点击 Advanced展开左侧菜单
2、点击蓝色按钮选择搜索字段在对应右侧输入框填写内容
3、如果不需要字段置空或者点击 Remove移除
4、填写如下内容即可搜索得到还有蛋白质三维结构的蛋白质列表
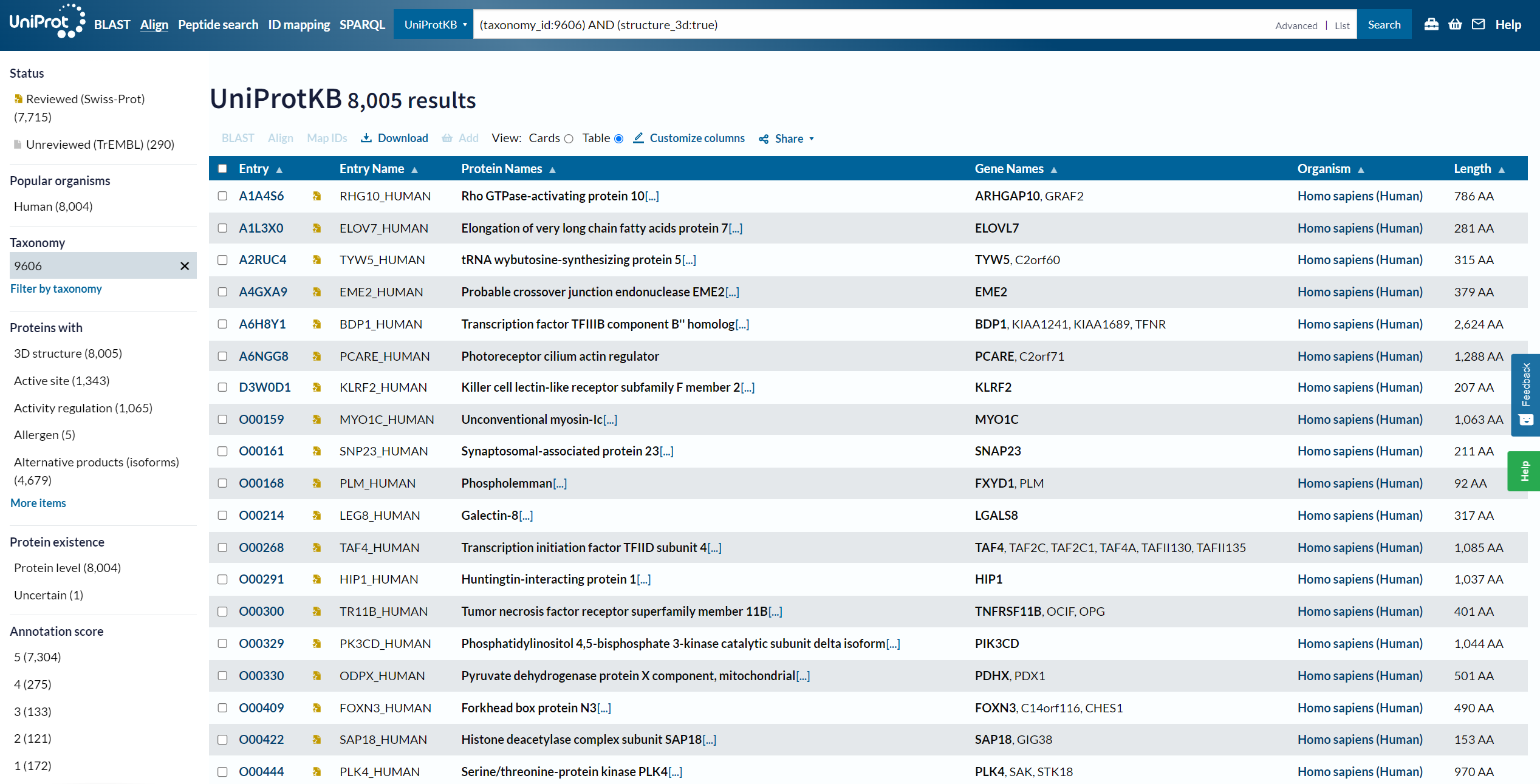
5、进入搜索结果
[https://www.uniprot.org/uniprotkb?query=(taxonomy_id:9606)%20AND%20(structure_3d:true)](https://www.uniprot.org/uniprotkb?query=(taxonomy_id:9606) AND (structure_3d:true))
3.4、蛋白质 ID 转换
1、进入官网https://www.uniprot.org/
可以在两个入口点击后进入

2、工作页面
这里以下列蛋白质 ID 作为输入搜索这些蛋白质三维结构的 PDB ID 为例
A1A4S6
A1L3X0
A2RUC4
A4GXA9
A6H8Y1
A6NGG8
D3W0D1
O00159
O00161
O00168
O00214
O00268
O00291
O00300
O00329
O00330
O00409
O00422
O00444
O00478
O00487
O00506
O00560
O00622
O00746
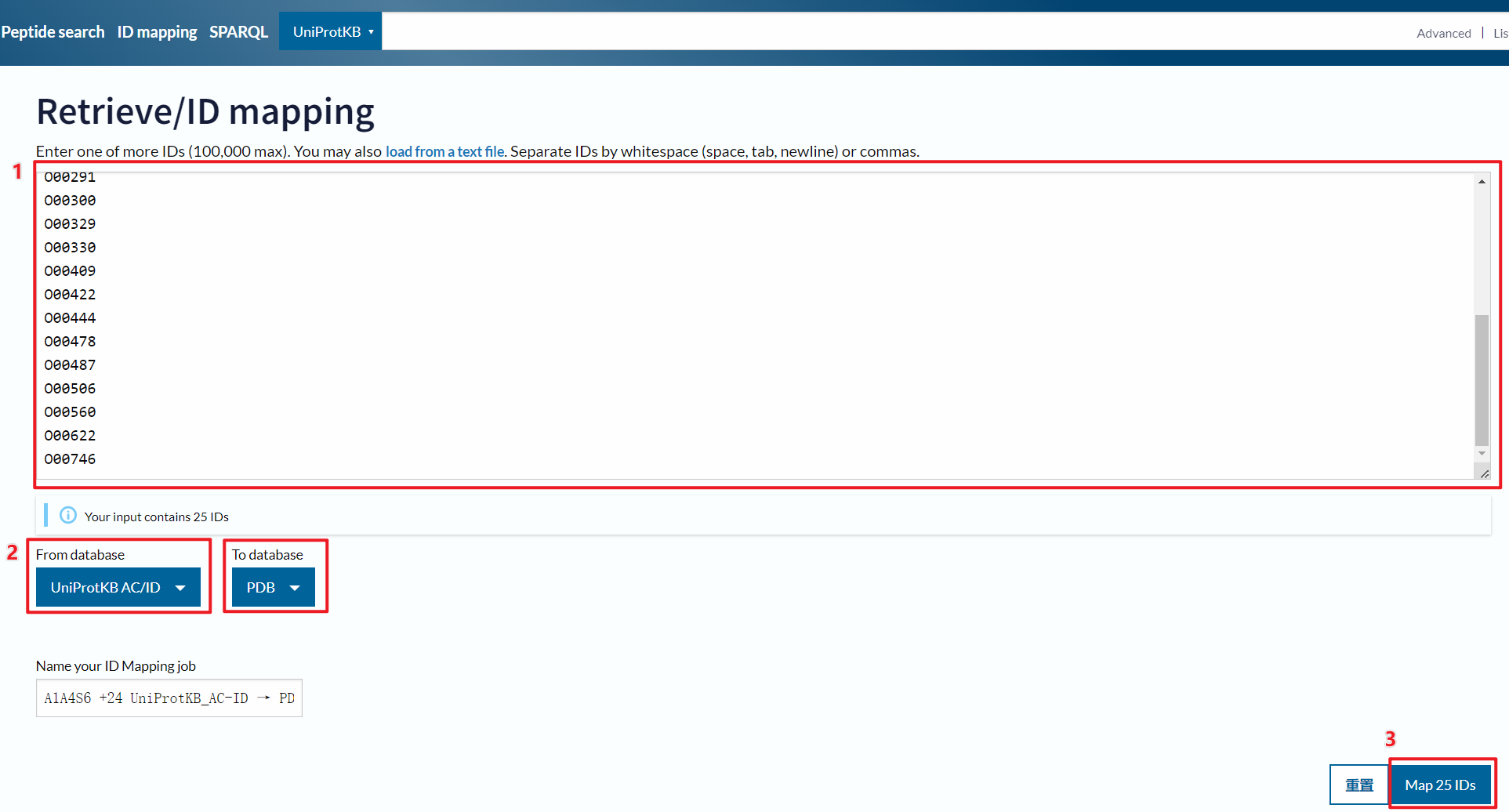
在 From database中选择我们的输入数据格式是 UniprotKB AC/ID
在 To database中选择我们需要转换的 PDB
点击 Map进入工作流程
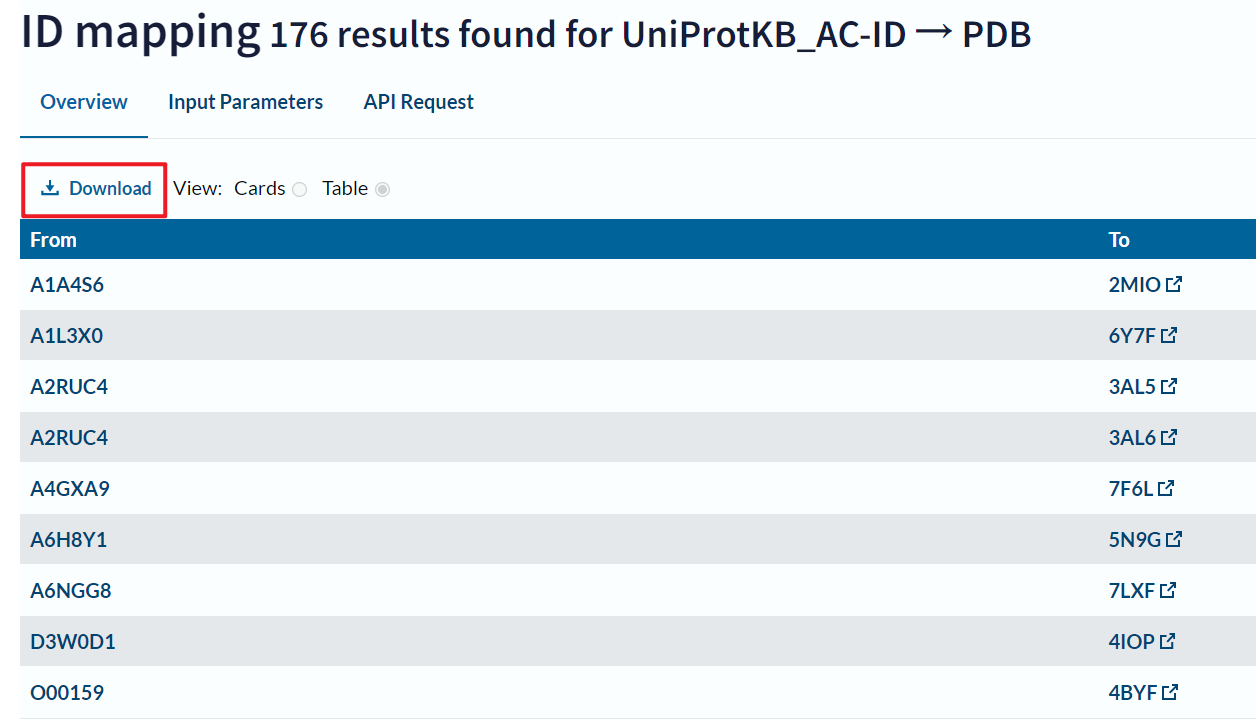
点击 Completed进入工作结果页面
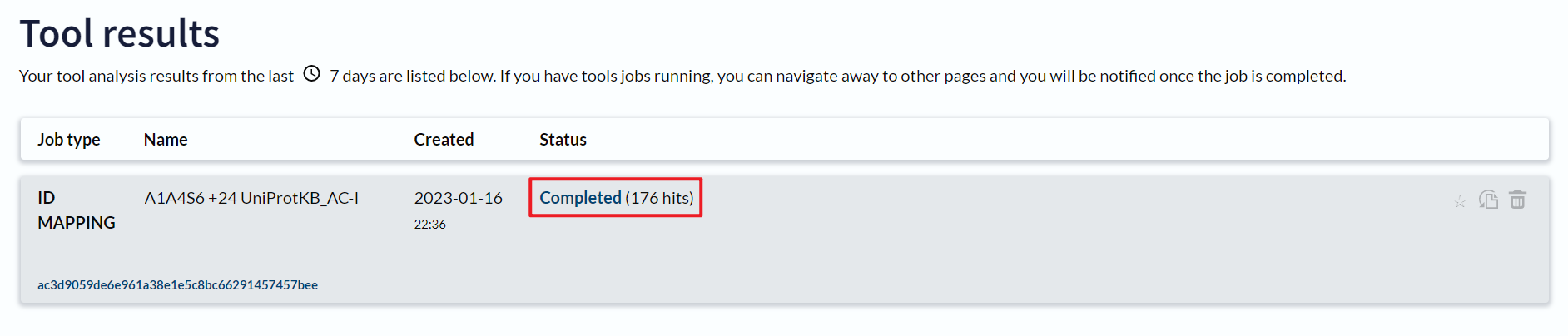
这里显示了匹配的 PDB id因为一个 蛋白质ID 可能对应多个PDB ID因此转换结果较多。
点击 Download进行下载
四、UniProtKB/TrEMBL 介绍
在认识到序列数据的生成速度超过了Swiss-Prot的注释能力时为了给不在Swiss-Prot中的那些蛋白质提供自动注释UniProt创建了TrEMBL翻译的EMBL核苷酸序列数据库。
在三大核酸数据库EMBL-Bank/GenBank/DDBJ中注释的编码序列都会被自动翻译并加入该数据库中。它也有来自PDB数据库的序列以及Ensembl、Refeq和CCDS基因预测的序列。之前提到的PIR组织制作了蛋白质序列数据库PIR-PSD也包含在其中。
五、UniParc 介绍
UniProt ArchiveUniParc包含来自主要公共可用蛋白质序列数据库的所有蛋白质序列的非冗余数据集。蛋白质可能存在于几个不同的来源数据库中并且在同一数据库中存在多个副本。 为了避免冗余UniParc仅将每个唯一序列存储一次。 相同序列被合并无论它们来自相同还是不同物种。 每个序列都有一个稳定且唯一的标识符UPI从而可以从不同的来源数据库中识别相同的蛋白质。
UniParc仅包含蛋白质序列没有注释。 UniParc条目中的数据库交叉引用允许从源数据库检索有关该蛋白质的更多信息。 当源数据库中的序列发生更改时UniParc将跟踪这些更改并记录所有更改的历史记录。
六、UniRef 介绍
UniProt Reference ClustersUniRef聚类序列可显著减小数据库大小从而加快序列搜索的速度。用于计算的蛋白质序列来自UniProtKB和部分UniParc记录的序列。
UniRef100序列将相同的序列和序列片段来自任何生物合并到一个UniRef条目中用于显示代表性蛋白质的序列。 使用CD-HIT算法对UniRef100序列进行聚类并构建UniRef90和UniRef50。UniRef90和UniRef50分别代表每个簇由与最长序列分别具有至少90%或50%序列同一性的序列组成。
七、蛋白质结构和序列预测的更新
最新版的 UniProt 整合了深度学习模型预测的结果包括
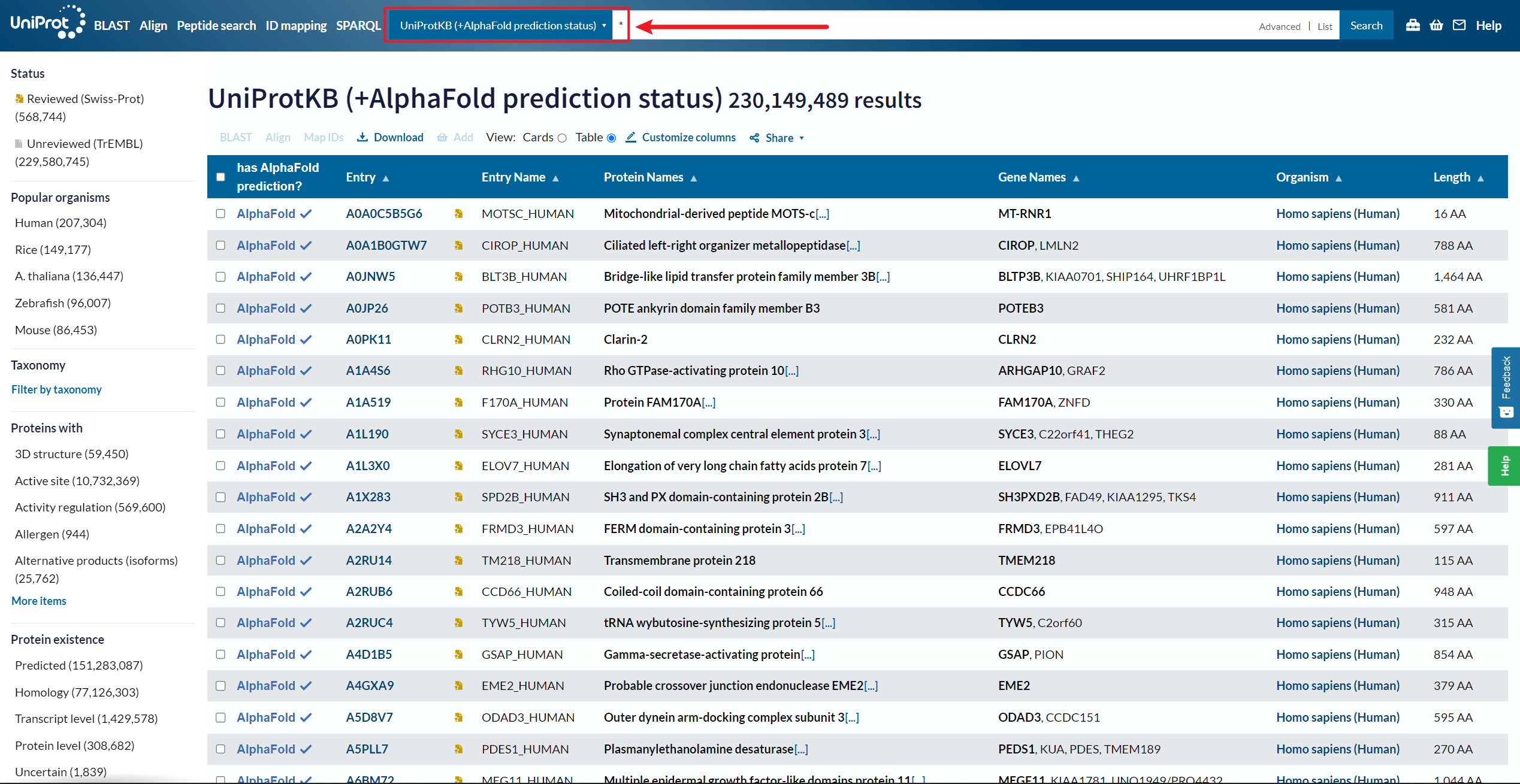
1、由 DeepMind 团队构建 AlphaFold2 预测的蛋白质三维结构
官网https://alphafold.ebi.ac.uk/
Uniprot 资源https://www.uniprot.org/alphafold?query=*
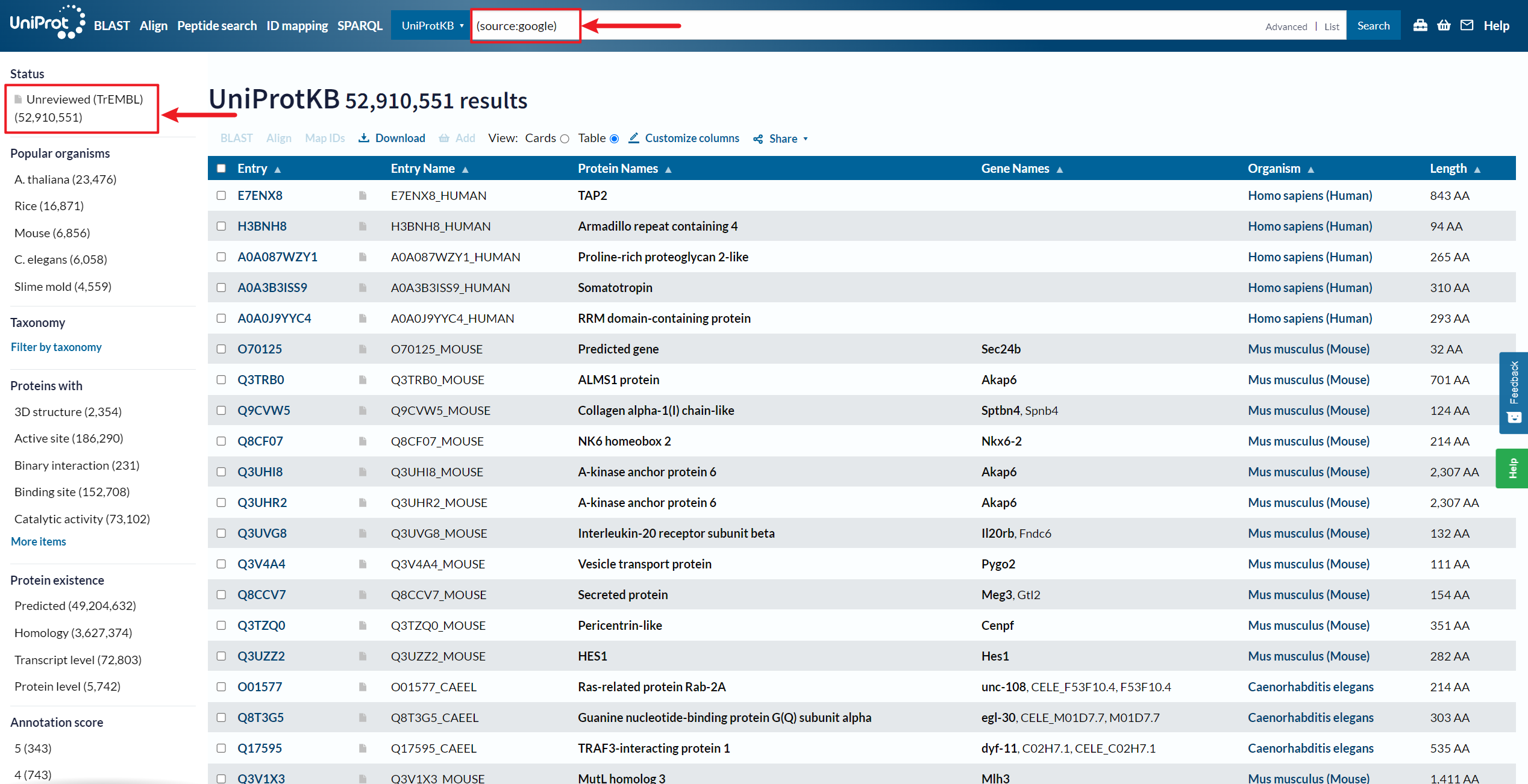
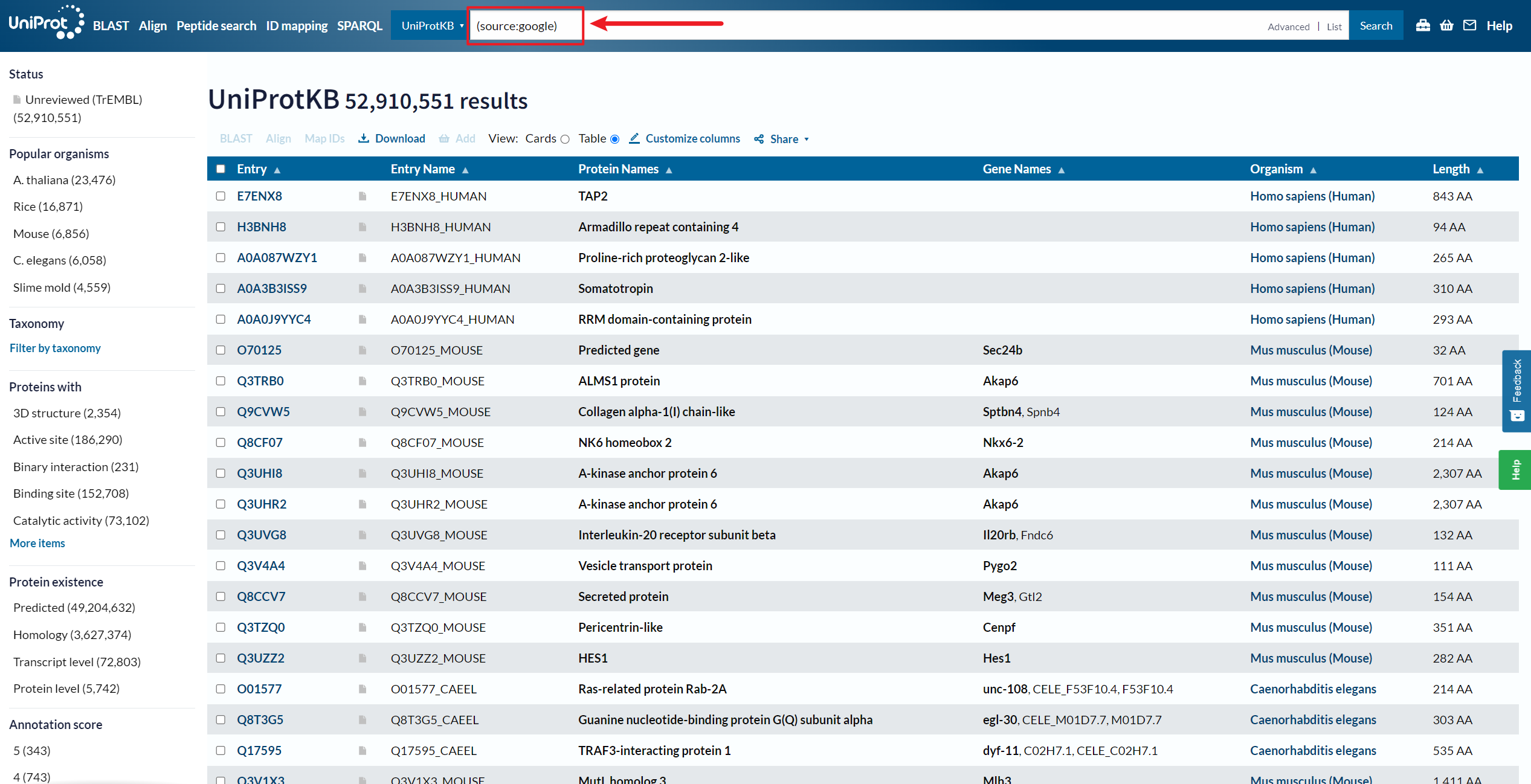
2、由 Google Brain 团队构建ProtNLM 预测的蛋白质序列注释
预印本https://proceedings.neurips.cc/paper/2017/file/3f5ee243547dee91fbd053c1c4a845aa-Paper.pdf
UniProt 资源https://www.uniprot.org/uniprotkb?query=(source:google)
不得不说现在生信是越来越卷了。。。
| 阿里云国内75折 回扣 微信号:monov8 |
| 阿里云国际,腾讯云国际,低至75折。AWS 93折 免费开户实名账号 代冲值 优惠多多 微信号:monov8 飞机:@monov6 |

